2.3 Leyendas¶
Graficar datos superpuestos y editar etiquetas y leyendas¶
Mira este video de 16:55 a 19:11
Para español, haga click en configuración, seleccione “español” debajo de los subtítulos.
Traducción por Evelia L. Coss-Navarrete, Langebio-Cinvestav, Irapuato, México)
# Para reproducir el siguiente tutorial, presiona shift + enter
from IPython.display import YouTubeVideo
from datetime import timedelta
start=int(timedelta(hours=0, minutes=16, seconds=55).total_seconds())
end=int(timedelta(hours=0, minutes=19, seconds=11).total_seconds())
YouTubeVideo("PJ1dvAgOAj0",start=start,end=end,width=640,height=360)
La siguiente es una transcripción del video.
💡 Recuerde: primero debe importar
matplotlibpara poder hacer gráficos. A continuación se muestran los datos con los que trabajaremos en esta lección que se utilizan en el video. Estas son listas de valores de coordenadasxeypara 15 especies diferentes de Vitis y Ampelopsis. Ejecute la celda a continuación para leer los datos para completar esta lección.
# Ejecute esta celda para importar matplotlib
import matplotlib.pyplot as plt
%matplotlib inline
# Esta celda contiene la lista de valores x y y para
# las formas de las hojas de las 15 especies de Vitis y Ampelopsis.
# Cada lista está abreviada con la inicial del género y epíteto de la especie.
# Ampelopsis acoutifolia (en realidad aconitifolia)
# NOTA: Hay un error de transcripción para esta especie, que en realidad es
# Ampelopsis aconitifolia (no es A. "acoutifolia")
# El error se propaga en los videos, narración, texto y cuadernos
# pero no afecta la lección en cuestión para trazar en matplotlib
Aaco_x = [13.81197507,-14.58128237,-135.3576208,-3.48017966,-285.0289837,-4.874351136,-126.9904669,10.54932685,170.4482865,40.82555888,205.158889,124.6343366,13.81197507]
Aaco_y = [27.83951365,148.6870909,157.2273013,35.73510131,-30.02915903,9.54075375,-280.2095191,0.200400495,-234.1044141,20.41991159,41.33121759,96.75084391,27.83951365]
# Ampelopsis brevipedunculata
# NOTA: Esta especie ahora se conoce como A. glandulosa var. brevipedunculata
Abre_x = [40.00325135,-81.37047548,-186.835592,-139.3272085,-287.5337006,-89.61277053,-134.9263008,47.43458846,144.6301719,163.5438321,225.9684307,204.719859,40.00325135]
Abre_y = [96.8926433,203.3273536,134.0172438,99.7070006,-81.35389923,-17.90701212,-335.624547,-80.02986776,-262.0385648,-27.31979918,-42.24377429,82.08218538,96.8926433]
# Ampelopsis cordata
Acor_x = [41.26484889,-99.68651819,-203.5550411,-181.4080156,-226.4063517,-174.1104713,-142.2197176,81.25359041,113.9079805,205.9930561,230.8000389,226.6914467,41.26484889]
Acor_y = [105.1580727,209.8514829,131.8410788,111.9833751,-70.79184424,-60.25829908,-326.5994491,-170.6003249,-223.0042176,-44.58524791,-45.80679706,71.64004113,105.1580727]
# Vitis acerifolia
Vace_x = [47.55748802,-102.1666218,-218.3415108,-183.5085694,-234.8755094,-152.1581487,-113.8943819,53.48770667,84.83899263,206.557697,240.589609,243.5717264,47.55748802]
Vace_y = [111.9982016,241.5287104,125.6905949,110.350904,-108.1932176,-74.67866027,-283.2678229,-161.1592736,-243.1116283,-54.52616737,-68.953011,95.74558526,111.9982016]
# Vitis aestivalis
Vaes_x = [34.13897003,-59.06591289,-192.0336456,-169.5476603,-261.8813454,-154.4511279,-132.6031657,56.04516606,119.9789735,205.0834004,246.928663,209.2801298,34.13897003]
Vaes_y = [80.26320349,227.2107718,155.0919347,123.2629647,-86.47992069,-70.12024178,-317.80585,-156.8388147,-247.9415158,-31.73423173,-28.37195726,120.2692722,80.26320349]
# Vitis amurensis
Vamu_x = [36.94310365,-63.29959989,-190.35653,-180.9243738,-255.6224889,-172.8141253,-123.8350652,60.05314983,113.598307,218.8144919,238.6851057,210.9383524,36.94310365]
Vamu_y = [87.06305005,230.9299013,148.431809,128.4087423,-88.67075769,-84.47396366,-298.5959647,-181.4317592,-241.2343437,-37.53203788,-30.63962885,115.7064075,87.06305005]
# Vitis cinerea
Vcin_x = [41.13786595,-78.14668163,-195.0747469,-185.81005,-238.1427795,-181.5728492,-127.6203541,65.24059352,103.8414516,214.1320626,233.1457326,222.7549456,41.13786595]
Vcin_y = [98.40296936,233.6652514,136.6641628,117.9719613,-86.41814245,-86.14771041,-310.2979998,-190.9232443,-230.5027809,-50.27050419,-42.94757891,107.8271097,98.40296936]
# Vitis coignetiae
Vcoi_x = [36.29348151,-51.46279315,-183.6256382,-176.7604659,-253.3454527,-191.8067468,-123.413666,66.11061054,111.4950714,215.7579824,236.7136632,197.5512918,36.29348151]
Vcoi_y = [86.42303732,222.7808161,150.0993737,127.4697835,-85.23634837,-93.3122815,-301.819185,-203.7840759,-239.8063423,-35.30522815,-25.15349577,121.1295308,86.42303732]
# Vitis labrusca
Vlab_x = [33.83997254,-63.35703212,-191.4861127,-184.3259869,-257.3706479,-179.056825,-124.0669143,68.23202857,123.213115,222.8908464,243.056641,205.2845683,33.83997254]
Vlab_y = [81.34077013,222.8158575,153.7885633,132.4995037,-80.2253417,-80.67586345,-296.8245229,-185.0516494,-238.8655248,-38.2316427,-29.21879919,111.424232,81.34077013]
# Vitis palmata
Vpal_x = [31.97986731,-68.77672824,-189.26295,-164.4563595,-260.2149738,-149.3150935,-131.5419837,65.86738801,127.3624336,202.6655429,240.0477009,219.0385121,31.97986731]
Vpal_y = [78.75737572,232.9714762,149.7873103,124.8439354,-71.09770423,-56.52814058,-329.0863141,-149.308084,-231.1263997,-33.22358667,-33.0517181,114.3110289,78.75737572]
# Vitis piasezkii
Vpia_x = [18.70342336,-28.68239983,-133.7834969,-32.76128224,-305.3467215,-7.429223951,-146.2207875,21.81934547,163.1265031,65.21695943,203.4902238,139.6214571,18.70342336]
Vpia_y = [41.05946323,160.3488167,157.9775135,64.93177072,-59.68750782,18.85909594,-362.1788431,7.556816875,-253.8796355,21.33965973,17.69878265,93.72614181,41.05946323]
# Vitis riparia
Vrip_x = [44.65674776,-85.47236587,-205.1031097,-174.088415,-239.9704675,-161.1277029,-125.4900046,58.08609552,89.2307808,204.9127104,236.0709257,229.8098573,44.65674776]
Vrip_y = [106.5948187,235.8791214,130.341464,116.8318515,-110.5506636,-76.73562488,-300.1092173,-169.0146383,-247.0956802,-42.2253331,-54.23469169,103.9732427,106.5948187]
# Vitis rupestris
Vrup_x = [51.29642881,-132.9650549,-227.6059714,-201.31783,-207.965755,-149.2265432,-98.64097334,48.33648281,75.91437502,208.7784453,237.4842778,263.3479415,51.29642881]
Vrup_y = [123.7557878,233.5830974,109.6847731,95.43848563,-95.82512925,-80.06286127,-236.7411071,-163.7331427,-213.2925544,-77.04510916,-86.40789274,69.86940263,123.7557878]
# Vitis thunbergii
Vthu_x = [22.61260382,-3.204532702,-150.3627277,-79.39836351,-271.8885204,-70.74704134,-168.6002498,36.68300146,172.978549,116.9174032,227.8346055,148.3453958,22.61260382]
Vthu_y = [50.82336098,194.3865012,181.2536906,86.8671412,-57.33457233,-23.85610668,-334.279317,-67.36542042,-234.1205595,7.151772223,28.16801823,138.9705667,50.82336098]
# Vitis vulpina
Vvul_x = [39.44771371,-83.62933643,-194.2000993,-175.9638941,-227.8323987,-180.8587446,-135.986247,71.94543538,99.8983207,207.0950158,231.7808734,222.7645396,39.44771371]
Vvul_y = [96.44934373,230.0148139,136.3702366,119.8017341,-83.09830126,-75.38247957,-332.9188424,-184.4324688,-222.8532423,-41.89574792,-44.70218529,101.9138055,96.44934373]
# Average grape leaf
avg_x = [35.60510804,-67.88314703,-186.9749654,-149.5049396,-254.2293735,-135.3520852,-130.4632741,54.4100207,120.7064692,180.696724,232.2550642,204.8782463,35.60510804]
avg_y = [84.95317026,215.7238025,143.85314,106.742536,-80.06000256,-57.00477464,-309.8290405,-137.6340316,-237.7960327,-31.10365842,-30.0828468,103.1501279,84.95317026]
Hablemos más de poner más de un conjunto de datos en un gráfico y cómo usar leyendas.
Entonces esto es muy fácil. Ya ha visto cómo puede usar múltiples funciones dentro de una gráfica, pero también puede llamar a plt.plot() varias veces con diferentes datos para darle diferentes atributos.
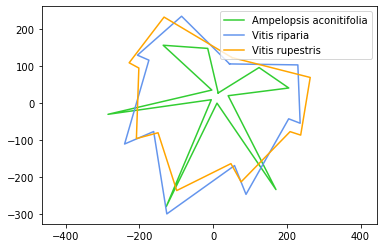
Entonces, aquí, por ejemplo, estamos usando plt.plot tres veces con tres especies de uva diferentes, esto es Ampelopsis aconitifolia, Vitis riparia y Vitis rupestris. Les estamos dando cada uno de un color diferente, pero lo que estamos haciendo aquí esta vez es que estamos usando un argumento llamado etiqueta. Así que le estamos dando
un nombre a estos datos en particular y este nombre es solo para esto datos. Puedes ver que es solo el nombre de la especie aquí. Es “Ampelopsis aconitifolia”, esta etiqueta es “Vitis riparia” para los datos de riparia, y esta etiqueta es “Vitis rupestris” para los datos de rupestris. Tenemos nuestro eje y la relación set_aspect para que
nuestra gráficasea proporcional.
Queremos ver nuestro eje, pero ahora tenemos una función adicional, plt.legend(), y estamos diciendo ubicación en la parte inferior se fue porque lo que va a hacer es tomar estas etiquetas y debido a que llamamos plt.plot() tres veces, gráfica los datos uno encima del otro. Pero debido a que tenemos esta etiqueta cuando le damos un nombre a cada uno de estos conjuntos de datos. ¿Qué sucede? Es cuando llamamos a plt.legend() podemos crear una leyenda y puedes elegir a dónde va. Correcto ahora está en la parte inferior izquierda. Podría decir abajo a la derecha, por ejemplo. Incluso tiene una opción llamada “best”, por lo que plt.legend() decidirá por sí mismo cuál cree que es la mejor ubicación para una leyenda.
# Use etiqueta para crear etiquetas para cada elemento de datos que desee mostrar en la leyenda
# Luego, use plt.legend () para crear una leyenda y colóquela
plt.plot(Aaco_x, Aaco_y, color="limegreen",label="Ampelopsis aconitifolia")
plt.plot(Vrip_x, Vrip_y, color="cornflowerblue", label="Vitis riparia")
plt.plot(Vrup_x, Vrup_y, color="orange", label="Vitis rupestris")
# Nota en el video se usa plt.axes.set_aspect ()
# Pero esto crea una advertencia, por lo que se usa gca () (Get Current Axes) en su lugar
plt.gca().set_aspect("equal", "datalim")
plt.axis("on")
plt.legend(loc="best") # o "best"
<matplotlib.legend.Legend at 0x7f1666f9f4c0>